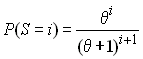- Etablissez le graphe de cette distribution pour le paramètre de mutation q calculé dans l'exercice précédent.
- Comparez cette distribution attendue avec la distribution mismatch
observée dans un échantillon de 20 gènes simulés par l'applet TreeToy
sur
http://www.xmission.com/~wooding/TreeToy/
Utilisez les paramètres suivants:- Sample size: 20
- Theta0: q calculé dans l'exercice précédent
- Growth Factor: 1.0
- Tau: 0 (ou n'importe quelle autre valeur)