| Cours I | Cours II | Cours III | Cours IV | Cours V | Cours VI |
|
Introduction |
Fréquences
alléliques Dérive génétique |
Introduction à la coalescence |
Changements démographiques |
Populations subdivisées |
Sélection Tests de neutralité |
| TP I | TP II | TP III | TP IV | TP V | TP VI |
Cours VI |
Tests de neutralité sélective |
Pour mettre en évidence des mécanismes de sélection au niveau génétique et moléculaire, il importe de définir auparavant ce que l'on attend en absence de sélection. Si les observations ne peuvent être expliquées sous un modèle d'évolution neutre, alors seulement on pourra invoquer l'action de la sélection.
On a donc paradoxalement besoin de définir des modèles d'évolution neutralistes avant d'étudier l'effet de la sélection. Il existe plusieurs modèles de sélection comme la sélection directionnelle, balancée ou diversifiante. Cependant, la simple compréhension de ces phénomènes sélectifs ne nous permet pas décider si le polymorphisme observé (p. ex. la distribution des fréquences alléliques) est compatible ou non avec la théorie neutraliste.
Plusieurs auteurs se sont donc attachés à définir la distribution de différentes quantités observables sous l'hypothèse neutraliste, afin de permettre de décider de la vraisemblance du modèle neutraliste. Il est aussi important de noter la plupart de ces dérivations ont été obtenues sous l'hypothèse supplémentaire de stationarité démographique de la population. Ces test de neutralités sont donc plus exactement des test de neutralité sélective et d'équilibre des populations. Un écart significatif à l'attendu pourra donc être dû à un phénomène de sélection ou à un écart à l'équilibre démographique de la population, comme à un bottleneck ou une expansion.
Warren Ewens a dérivé en 1972 la distribution attendue des fréquences de k allèles dans un échantillon de taille n. Sans entrer dans les détails de la dérivation, il a montré que cette distribution conditionnée par le nombre d'allèles observés (k) était indépendante du paramètre de mutation de la population q = 4Nu. Cette distribution s'obtient comme
où les ni sont les nombres de gènes
du type allélique i, et n est le nombre total de
gènes dans l'échantillon. la taille de l'échantillon, et ![]() est
un nombre de Stirling du premier genre, c'est à dire le
coefficient devant q k
de l'expansion
est
un nombre de Stirling du premier genre, c'est à dire le
coefficient devant q k
de l'expansion ![]() .
.
Cette distribution permet d'obtenir la distribution attendue des fréquences alléliques dans une population. Stewart (1977) a décrit un algorithme (implanté dans Arlequin) permettant d'obtenir des échantillons aléatoires tirés de cette distribution, et par la même la distribution attendue des fréquences alléliques ou la distribution de toute autre quantité basée sur les fréquences alléliques.
Distribution de fréquences d'alléles RFLP dans 2 populations humaines |
|
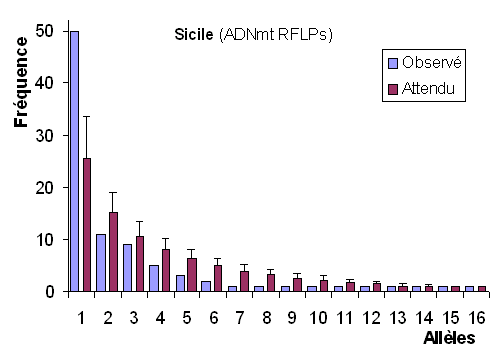 |
L'allèle observé le plus
fréquent est beaucoup plus fréquent que ce que
l'on attend sous l'hypothèse neutraliste pour une
population stationaire. Il y a aussi un défaut d'allèles présentant des fréquences intermédiaires. Il y a donc un écart significatif entre les distributions observées et attendues. |
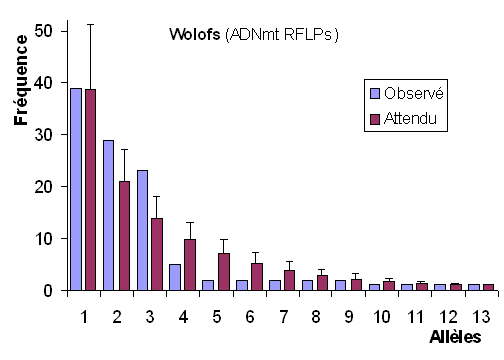 |
Les 2 distributions sont ici par contre en assez bon accord. L'hypothèse neutraliste peut être acceptée. |
Ewens (1972) a aussi établi que la probabilité d'observer k types alléliques dans un échantillon de taille n était donné par
A partir de la dernière relation, on peut obtenir l'espérance du nombre d'allèles dans un échantillon de taille n comme
Nombre d'allèles attendus dans un échantillon de taille arbitraire pour différentes valeurs du paramètre de mutation q
Cette relation permet de prédire le nombre d'allèles que l'on observerait pour d'autres tailles d'échantillons que celle que l'on observe. Ceci est utile pour 2 raisons:
Il y abien sûr une relation étroite entre la théorie de l'échantillonnage de Ewens et la théorie de la coalescence avec mutation. Les simulations par coalescence peuvent d'ailleurs permettre d'obtenir des échantillons aléatoires de taille donnée possédant un certain nombre d'allèles. En ne gardant que les échantillons ayant le nombre d'allèle observé, on obtiendra la même distribution que celle donnée par la formule de Ewens.
Bien que des résultats théoriques existent concernant l'espérance et la variance de différentes statistiques portant sur la diversité moléculaire neutre dans une population stationnaire (comme le nombre de sites polymorphes, l'homozygotie, ou le nombre moyen de différences par paires), la théorie de la coalescence permet d'obtenir facilement et rapidement toute la distribution de ces statistiques. C'est pour cela que des simulations basées sur la théorie de la coalescence sont à la base même de la plupart des tests actuels de neutralité sélective.
L'évolution des fréquences alléliques sous l'effet de la la sélection directionnelle ressemble fortement à l'évolution d'une population en croissance logistique.
Croissance logistique d'une population
Evolution de la fréquence de l'allèle A1
sous l'effet de la sélection directionnelle
La généalogie des gènes porteurs de l'allèle A1 peut donc fortement ressembler à celle de gènes trouvés dans une population ayant connu une expansion récente (Slatkin et Hudson 1991), c'est à dire à une généalogie en étoile (star-shape genealogy) ou en peigne, selon la façon dont on dessine ces généalogies. On note cependant qu'une telle observation n'est possible que si la fitness absolue des porteurs de l'allèle favorablement sélectionné augmente aussi. Il faut donc que le nombre de gènes de type A1 augmente fortement dans la population. Dans une population de grande taille, on aura donc assez facilement des généalogies en étoile après un épisode de sélection directionnelle. Cependant, dans des populations de petite taille, la sélection directionnelle conduit le plus souvent à un balayage sélectif (selective sweep) du polymorphisme, avec une généalogie en étoile de trop faible taille pour voir l'apparition de beaucoup de mutations. La fixation de la nouvelle mutation va donc conduire à y effacer la diversité génétique préexistante.
Dans le cas de la sélection balancée, les allèles vont avoir tendance à être conservés pendant une plus grande période dans la population que pour des allèles neutres. Dans la figue ci-dessous, on voit qu'un allèle se maintient dans une population de taille finie pendant une très longue période, alors que des mutations neutres se fixent beaucoup plus rapidement .
Succession de fixations de mutation neutres Polymorphisme maintenu par sélection balancée
pendant une longue période
Les généalogie de tels gènes devraient donc ressembler schématiquement à ceci:
Diversité moléculaire accumulée sur une généalogie de gènes neutres Diversité moléculaire accumulée sur une généalogie de gènes maintenus par sélection balancée (avantage d'un hétérozygote)
On s'attend à ce que la généalogie de gènes soumis à sélection balancée soit globalement plus longue qu'une généalogie de gènes neutres, et donc qu'un locus soumis à sélection balancée maintienne plus de variabilité qu'un locus neutre.
Kreitman et Hudson (1991) ont analysé le polymorphisme de séquence de la région du gène Adh (environ 5Kb), et ils ont noté un fort excès de polymorphisme autour d'un site polymorphe responsable de l'occurrnce de deux variants électrophorétiques (fast: F et slow: S). Ce polymorphisme semble être soumis à sélection balancée par un mécanisme d'avantage des hétérozygotes. On retrouve aussi un gradient de fréquence de l'allèle F en fonction de la latitude, qui semble être plus avantagé dans les régions froides, vraisemblablement grâce à une meilleure activité enzymatique à basse température, pemettant aux mouche de mieux transformer les alcools en sucre. L'allèle S a d'autre part une variabilité associée plus élevée que l'allèle F et semble donc plus ancien (Kreitman 1983).
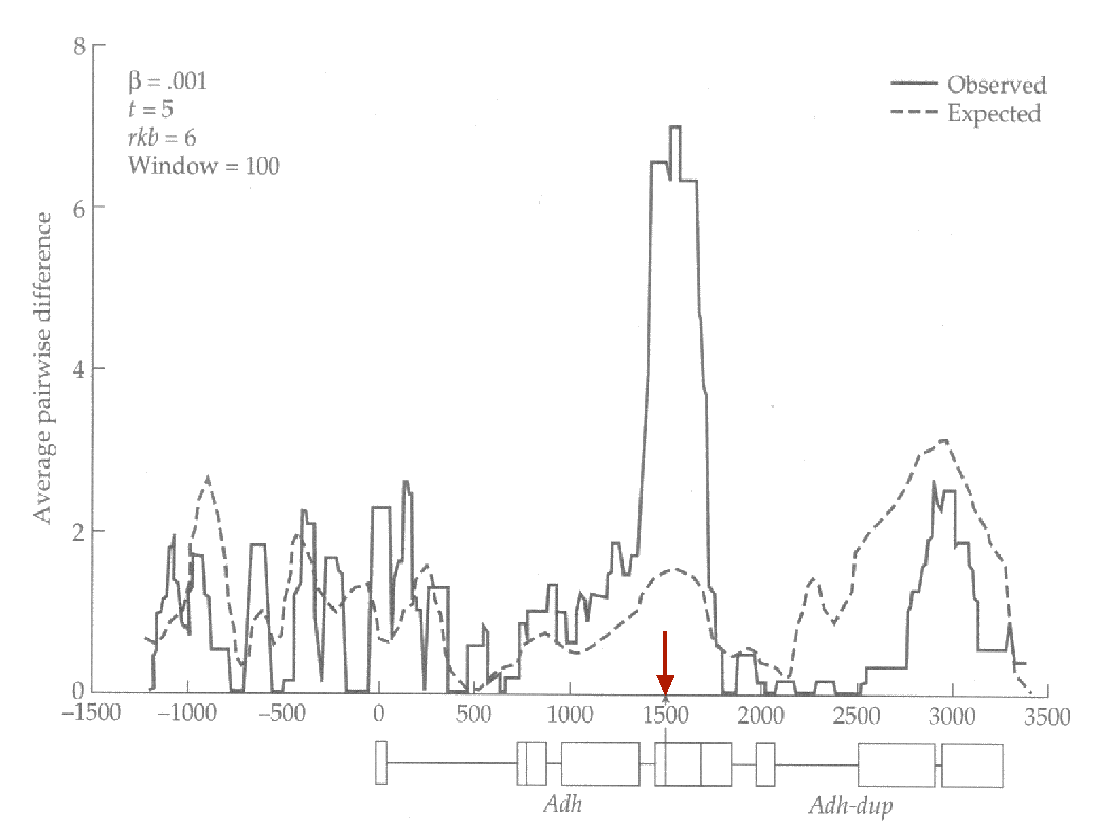 |
Diversité nucléotidique observée et attendue sous un modèle neutre dans la région du gène Adh de la drosophile. |
Sur cette figure, le pic de variabilité autour du site F/S semble bien être dû au polymorphisme balancé. La diversité génétique diminue rapidement lorsque l'on s'éloigne du site sélectionné à cause de la recombinaison. L'effet de la sélection balancée ne se fait donc sentir que pour quelques centaines de paires de bases autour du site sélectionné et pas au delà.
On peut donc imaginer qu'un screening de la diversité moléculaire dans des régions codantes pourrait mettre en évidence d'autres sites soumis à sélection balancée. Ceci n'a cependant pas été étudié pour d'autres locus qu'Adh. Dommage.
La sélection d'arrière plan est une mécanisme de sélection qui élimine des allèles (faiblement) désavantageux pour leur porteur (Charlesworth et al. 1993).
| Généalogie de gènes neutres | Généalogie de gènes avec sélection d'arrière plan (les lignages éliminés par la sélection sont en pointillé) |
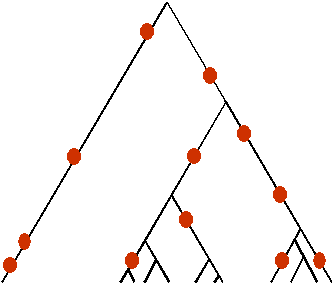 |
|
On constate que la sélection d'arrière plan conduit à une diversité réduite par rapport à la diversité attendue pour une gènealogie non soumise à sélection. Cependant, la forme de la généalogie est très semblable à celle d'une généalogie neutre et il est très difficile de distinguer l'action de la sélection d'arrière plan d'un taux de mutation réduit, ou encore d'un mécanisme de sélection directionnelle avec balayage sélectif (selective sweep) à un locus proche dont l'effet atténué se fait sentir par autostop (hitchhiking effect) au site étudié (Charlesworth et al. 1993).
Dans ce modèle de sélection, les mutations avantageuses se fixent rapidement et les allèles nuisibles à leur porteur sont éliminés. On s'attend à observer une diversité moléculaire réduite par rapport à un polymorphisme neutre.
Une publication récent a comparé la diversité moléculaire du gène du récepteur 1 de la mélanocortine MC1R (un gène qui explique une part importante de la variabilité de la pigmentation chez l'homme) en Europe et en Asie. Contrairement à beaucoup d'autres polymorphismes, on trouve beaucoup plus de diversité moléculaire en Europe qu'en'Afrique.
Phylogénies d'allèles du récepteur 1 de la mélanocortine en Europe (a) et en Afrique (b)
On remarque que toutes les mutations trouvées en Afrique sont synonymes, alors que nombre de mutations observées en Europe ne le sont pas. Malgré cela, le polymorphisme Européen est compatible avec un polymorphisme neutre, suggérant une relaxation des contraintes fonctionnelles pour ce gène en Europe par rapport à l'Afrique.
Watterson (1978, 1986) s'est basé sur la théorie d'échantillonnage de Ewens pour proposer un test de neutralité sélective basé sur l'homozygotie attendue d'un échantillon. Cette homozygotie attendue dépend uniquement des fréquence alléliques comme
Watterson a montré que cette statistique était une statistique suffisante pour rendre compte de la diversité génétique de l'échantillon. En pratique, test consiste à comparer la valeur de la statistique F à celles obtenues à partir d'échantillon simulés sous l'hypothèse de neutralité et de stationarité de la population. On utilise le plus souvent un algorithme décrit par Stewart (1977) pour générer des échantillons aléatoires de même taille et possédant le même nombre d'allèles que l'échantillon observé. Pour chaque échantillon simulé, on calcule la statistique F, et l'on obtient empiriquement la probabilité associée à F comme la fraction des échantillons donnant une valeur de F inférieure ou égale à celle observée.
Slatkin a proposé d'utiliser directement la probabilité de l'échantillon observé au lieu d'utiliser la statistique F pour batir un test exact de neutralité sur la base des fréquences alléliques. Le principe en est le même et donne des résultats le plus souvent comparables à ceux du test de Ewens-Watterson. On peut énumérer exhaustivement outes les configuration alléliques possible et calculer la probabilité d'obsever un échantillon plus improbable que celui que l'on observe, en sommant directement les probabilités associées aux configurations plus improbables que celle observée. Alternativement, on peut simuler un grand nombre d'échantillon et estimer cette probabilité par la fraction des échantillons simulés étant plus improbables que l'échantillon observé.
Ces 2 procédures sont implémentées dans le logiciel Arlequin.
Un processus de sélection directionnelle, mais aussi une croissance récente de la population vont conduire à un l'observation d'un allèle très fréquent et d'un grand nombre d'allèles rares, et donc à un une valeur de F trop élevée par rapport à celle attendue pour un gène neutre ou une population stationaire. Cette forme d'écart est par exemple trouvée pour les fréquences alléliques de l'ADN mitochondrial (voir le cas de la population de Sicile mentionné plus haut).
A l'inverse, une forme de sélection balancée devrait conduire à observer des fréquences alléliques trop égales et donc à une valeur de F trop faible. C'est effectivement ce que l'on trouve pour le système HLA.
Tajima (1983) a proposé un des premiers test de neutralité sélective basé sur la diversité moléculaire des échantillons.le principe de ce test est de comparer l'estimation du paramètre de mutation q = 4Nu obtenue à partir du nombre de sites polymorphes S (qS) à celle obtenue à partir du nombre moyen de différences entre 2 gènes p, qui est précisément une estimation de qp . Tajima a ainsi défini la statistique D comme
avec ![]() = p et
= p et ![]() comme étant égale à
comme étant égale à
que nous avons déja vue. Le dénominateur est une expression compliquée que nous ne développerons pas. Comme dans le cas du test de Ewens-Watterson, la valeur observée de D est comparée à celles obtenues en simulant des échantillons tirés d'une population stationnaire de paramètre de mutation qS . Dans le logiciel Arlequin, on utilise des simulations du processus de coalescence pour générer ces échantillons.
Ce test va dépendre de la différence de comportement de qp et qS dans différentes situations.
Sélection directionnelle et purificatrice: S est peu affecté par la forme de la génalogie mais beaucoup par sa longueur totale. Par contre p est affecté par les 2 facteurs, et sa valeur dépendra avant tout de la diversité des allèles les plus fréquents. Après un épisode de balayage sélectif, on aura beaucoup d'alèles rares qui contribueront peu à p mais beaucoup à S, si bien que l'on s'attend à avoir des valeurs négatives de D.
Sélection balancée: Dans ce cas on aura un effet inverse, car des allèles de fréquences intermédiaires auront beaucoup d'effet sur p mais relativement peu sur S. D sera donc positif.
Expansion de population: On s'attend à avoir des valeurs fortement négatives de D, car le nombre de sites polymorphes croîtra relativement rapidement, alors qu'il y aura un excès d'allèles de faibles fréquences qui auront peu d'influence sur p.
Contraction de population: Après une contraction de population, le nombre de sites polymorphes diminue d'autant d'autant plus vite que la taille de l'échantillon est grand (Tajima, 1990). On s'attend donc à avoir des valeurs positives de D.
Bottleneck (contraction et réexpansion): Dans ce cas, S est initialement affecté plus fortement pour de grandes taille d'échantillon, mais la valeur d'équilibre est retrouvée plus rapidement qu'avec de petites tailles d'échantillon (n=2). p est affecté plus fortement que S . On s'attendra donc à observer initalement un D positif, puis ensuite un D négatif.
Evolution du D de Tajima pendant et après un bottleneck (Fay et Wu 1999)
Effet de l'hétérogénéité des taux de mutation sur D: Un taux hétérogène de mutation le long d'une séquence d'ADN a pour conséquence l'accumulation de plusieurs mutations au même site (un hot-spot de mutation). Cela a comme effet de produire une valeur de D positive, car le nombre de sites polymorphe S sera considérablement réduit par rapport au modèle des sites infinis, à cause de la présence de ces hot-spots. D'un autre côté, l'hétérogéité aura relativement peu d'effet sur p, car même si des mutations se produisent plusieurs fois au même site, elles vont le plus souvent s'inscrire dans un contexte de séquence différent des mutatins précédentes. On a aussi remarqué (Aris-Brosou et Excoffier, 1995) qu'en cas d'hétérogénéité du taux de mutation et d'une expansion de population, il était très dificile de pouvoir rejetter l'hypothèse neutraliste-stationarité du fait des effets antagonistes de ces 2 facteurs, qui conduit à de valeurs de D faiblement négatives.
Statistique D de Tajima dans des populations en expansion et avec présence de sites hot-spots de mutation. La distribution de D se trouve alors entièrement à l'intérieur de l'intervalle de confiance neutraliste.
Test basé sur la statistique FS (Fu 1997)
Ce test est un peu analogue au test de Tajima, mais au lieu de regarder si le nombre de différence par paires ( p ) est compatible avec le nombre de sites polymorphes, il se base sur la relation attendue entre p et le nombre d'allèles de l'échantillon (k). L'estimateur qp est utilisé pour calculer la probabilité d'observer k ou plus allèles dans l'échantillon au moyen de la formule de Ewens que nous avons vue plus haut. Fu défini ainsi la quantité
Dans un échantillon ayant un excès de nouvelles mutations, q estimé par qp devrait être plus petit que q estimé par le nombre d'allèle, et donc S ' devrait être un bon indicateur de la présence de mutation nouvelles. Pour éviter d'avoir des valeur critiques de S ' trop proches de zéro, Fu prend comme statistique le logit de S ', soit
FS aura tendance a être négatif si il y a un excès de mutations récentes (d'allèles rares).
Fu (1997) a montré que ce test était paticulièrement sensible pour détecter des expansions récentes (et donc aussi de la sélection directionnelle). La probabilité associée à la statistique F est obtenue par une série de simulation de processus de coalescence en prenant qp comme paramètre de mutation.
Test basé sur la statistique F (Fu et Li 1993)
Fu et Li (1993) ont développé plusieurs statistiques portant sur un autre aspect du polymorphisme présent dans une généalogie de gènes. Ils ont fait la distinction entre les mutations se produisant sur les branches externes et les branches internes d'une généalogie. Sous le modèle des sites infinis, on peut donc décomposer le nombre total de sites polymorphes entre ceux qui sont survenus dans les branches internes de la généalogies (des mutations anciennes) et ceux survenus sur des branches externes de la généalogies (des mutations récentes), comme
On note que he correspond aux nomre de mutations singletons de l'échantillon. Ils ont ensuite démontré que l'espérance de he était égal à
Comme on a vu que
On en déduit que
Normalement, p est beaucoup moins sensible que he à la présence de nouvelles mutations, et Fu et Li ont donc proposé d'utiliser la statistique
pour mettre en évidence ces nouvelles mutations.
D'autre part, en cas de sélection d'arrière plan, les mutations sur les branches internes de la généalogie seront certainement neutres alors que les mutation faiblement délétères seront plutôt présentes sur les branches externes si elles ne sont pas éliminées par la sélection. Fu et Li ont ainsi construit une autre statistique G pour mettre en évidence la présence de mutations faiblement délétères,
Par simulation, Fu a montré que les statistiques F et G était les plus puissantes pour détecter un phénomène de sélection d'arrière plan, car elle contrastaient les mutations singletons et non-singletons.
Il est donc intéressant de comparer les résultats de différents test pour essayer de mettre en évidence non seulement un écart à la neutralité, mais encore un modèle possible de sélection.
Ce TP est accessible sur gmdp_tp6.htm
Laurent Excoffier : Dernier update : mardi, 30 mai 2006 11:18